
产品货号:
YTB4150
中文名称:
基因编辑突变检测试剂盒
英文名称:
Gene-Editing Mutation Detection Kit
产品规格:
25T|100T
发货周期:
1~3天
产品价格:
询价
本制品是一种简单、可靠、快速的基于T7 Endonuclease I(T7EI)的用于检测基因组DNA在基因编辑后发生突变的试剂盒。本试剂盒主要利用了T7 Endonuclease I(T7EI)能识别并酶切不完全配对的DNA双链(也称异源双链DNA)的特性。本试剂盒提供了一步法基因组DNA提取试剂,仅需一步孵育就可完成基因组DNA的提取。
本试剂盒包含了一组对照模板和引物预混液,便于用作阳性对照。对照模板包含了未突变的和突变的基因片段,未突变的基因片段PCR扩增后的长度为525bp,突变的基因片段含有7个碱基的缺失突变,PCR产物为518bp。对照模板经PCR扩增、变性和退火处理后,会形成不完全配对的双链DNA,被T7EI酶切消化后,518bp的片段会被酶切成288bp和230bp两个不同大小的片段,而完全配对的双链DNA不会被T7EI酶切消化。这样通过凝胶电泳就可以非常清晰地观察到没有被剪切的525bp和518bp的条带,和被剪切开的288bp和230bp条带。通常525bp和518bp的条带由于分子量非常接近而仅呈现一个条带。
本试剂盒提供了包含了高保真酶BalbFusion DNA Polymerase的BalbFusion Master Mix,确保扩增产物能被高保真扩增,不会产生基因组编辑突变检测的假阳性。
本试剂盒的PCR扩增产物可以直接用于变性退火和T7EI进行酶切。本试剂盒的PCR扩增体系和T7EI酶兼容,PCR结束后无需进行PCR产物的纯化,直接进行变性退火后就可以加入T7EI酶进行酶切鉴定分析,并且不完全匹配的双链DNA仅需15分钟就可以被充分酶切消化。

首先需要编辑的细胞、组织或器官通过转染、病毒感染等技术手段表达CRISPR/Cas9、TALEN或ZFN等核酸酶。在细胞修复机制的介入下,这些核酸酶会在guide RNA(sgRNA)等的特异性指引下,在基因组的特异位点上产生插入或缺失突变(insertions or deletions,indels)。提取相应的基因组DNA后,通过高保真PCR扩增酶从被编辑的基因组DNA中扩增出被编辑过的DNA片段。扩增的目的片段进行变性和退火处理,然后用T7EI进行酶切鉴定。如果部分细胞中成功出现了基因编辑导致的插入或缺失突变,PCR扩增产物变性和退火后,未突变的和突变的基因之间,不同突变的基因之间,都会形成不完全配对的双链DNA。而T7EI酶能识别并酶切不完全配对的双链DNA。这样对于T7EI酶切产物进行电泳分析,就能知道基因组是否被成功编辑,以及被编辑的效率。

- 本试剂盒组分特别齐全:提供了包括一步法基因组DNA提取、高保真PCR扩增、变性退火和T7EI酶切的全套试剂,并且还提供了阳性对照。
- 本试剂盒使用特别便捷:一步法基因组DNA提取非常快速便捷,并且提取后可以直接用于高保真PCR扩增,PCR扩增产物也可以直接用于后续的变性退火和T7EI酶切。无需额外的分离纯化步骤。

| 组分 | 25T | 100T |
| DNA Extraction Solution | 1.2mL | 5mL |
| Enzyme Mix | 100μL | 400μL |
| 2×BalbFusion Master Mix | 800μL | 1.6mL×2 |
| T7 Endonuclease I | 25μL | 100μL |
| 10×Detection Reaction Buffer | 100μL | 400μL |
| Control Templates & Primers | 20μL | 20μL |
| 无核酸酶超纯水 | 200μL | 800μL |
保存:-20℃,避免反复冻融,有效期1年。

- 配制细胞裂解液时,DNA Extraction Solution和Enzyme Mix混匀后,应尽快使用,放置太久可能会影响DNA抽提效果。
- 对于首次使用本试剂盒的情况,强烈推荐使用本试剂盒提供的Control Templates & Primers作为阳性对照。其PCR产物经变性退火后,可以作为T7 Endonuclease I酶消化的阳性对照。

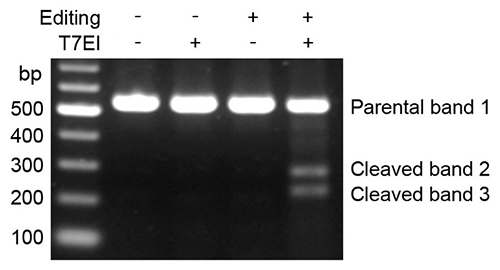
图1.本试剂盒检测HEK293T细胞基因组编辑突变的效果图。将HEK293T细胞种于六孔板中,待细胞融合度约80%的时候,用LP8000转染试剂将表达Cas9蛋白和靶向人Hace-1基因的guide RNA的质粒转染HEK293T细胞,同时转染空载质粒(empty vector)作为阴性对照。转染24h后消化细胞接种于96孔板中,每孔约3万个细胞,24h后,弃上清向孔内加入添加了Enzyme Mix的DNA Extraction Solution提取基因组(68℃,15min;95℃,10min)。用2×BalbFusion Master Mix扩增靶基因,扩增产物经变性和退火处理后(95℃ 5min;95℃~85℃,2℃/s;85℃~25℃,0.1℃/s),加入1μL T7EI,37℃酶切15min,2%琼脂糖凝胶电泳检测。转染含有靶向Hace-1基因的guide RNA的质粒时会形成插入或缺失突变,进行基因扩增后,未突变的和突变的基因片段配对会形成不完全匹配的双链DNA,被T7EI识别和酶切后,约520bp的退火片段中的不完全匹配的双链DNA会被酶切成约290bp和230bp的两个片段。转染空载质粒对靶基因没有影响,不会产生T7EI酶切产生的小片段。

- 引物设计:引物长度在18~22bp左右,GC含量在45%~60%之间,Tm>55℃,引物扩增的片段长度在500~1000bp之间,且突变位点最好不要在扩增片段正中央,以便酶切形成两个不同大小的片段便于进行电泳观察。
- 样品准备:对于细胞样品,收集约5~100万个细胞(不推荐超过200万个细胞)沉淀至PCR管中,或者如果经基因编辑的细胞接种在96孔板中,待细胞长至80~100%融合度时吸除培养液后备用。对于组织样品,总量约为1~5mg,不建议超过10mg,放至PCR管中。
- 基因组DNA提取:
- 消化液的配制。根据样本数量参考下表配制消化液,并充分混匀。
成分 1 Sample 10 Samples DNA Extraction Solution 48μL 480μL Enzyme Mix 2μL 20μL - 裂解液需现配现用,并充分混匀后使用。
- 基因组DNA的提取。每个样品加入50μL消化液。对于细胞沉淀,适当充分重悬;对于培养在96孔板中的细胞,作用1min后,适当吹打并全部转移至PCR管中;对于组织样品,可以适当反复吹打数次。按照以下条件在PCR仪或水浴锅(优先推荐使用PCR仪)进行孵育反应。
温度 时间 68℃ 15min 95℃ 10min 4℃ hold - 反应结束后立即进行后续的PCR扩增或在-20℃保存。
- PCR扩增:首次扩增建议参考下表设置不同的实验组以确保实验的顺利进行。
Group Template Sample Genomic DNA from Cells with Gene Editing Negative Control Genomic DNA from Cells without Gene Editing Positive Control Template Provided by this Kit Blank Control Water - PCR反应体系如下。
成分 样品 阳性对照 空白对照 基因组DNA 1μL - - Primer Mix(10μM each) 0.5μL - 0.5μL Control Templates & Primers - 0.5μL - 2×BalbFusion Master Mix 10μL 10μL 10μL 无核酸酶水 8.5μL 9.5μL 9.5μL 总体积 20μL 20μL 20μL - 用移液器轻轻吹打混匀或轻微Vortex混匀,室温离心数秒,使液体积聚于管底。
- 把配制好的PCR反应体系置于PCR仪上,开始PCR反应。
- PCR反应参数的设置如下。
步骤 温度 时间 循环数 预变性 95℃ 3min 1 变性 95℃ 20sec 35 退火 55℃ 30sec 延伸 72℃ 30sec 终延伸 72℃ 5min 1 Hold 4℃ - - - 选做:PCR结束后,每样取少量PCR产物用琼脂糖凝胶电泳检测目的条带是否单一和大小是否正确,如果正确则进行下一步实验。如果前期做过预实验,对于PCR扩增比较有把握,可以忽略本步骤。
- 消化液的配制。根据样本数量参考下表配制消化液,并充分混匀。
- 变性与退火:
- 按下表配制变性与退火体系。
成分 用量 PCR产物 5μL 10×Reaction Buffer 2μL 无核酸酶水 12μL 总体积 19μL - 扩增的PCR产物无需进一步纯化即可用于变性与退火。每个变性与退火反应所使用的PCR产物的量宜在50~250ng范围内,PCR产物用量过少,会导致后续电泳条带偏弱;PCR产物用量过多,会导致后续T7EI酶切不太充分。
- 按照下表的步骤,在PCR仪上进行变性和退火。
步骤 温度 时间 变性 95℃ 5min 退火 95℃~85℃ -2℃/sec 85℃~25℃ -0.1℃/sec Hold 4℃ -
- 按下表配制变性与退火体系。
- T7EI酶切鉴定:
- 在变性和退火完成后,每管加入1μL的T7EI。
- 37℃酶切15min。如果把酶切时间延长至15~60min也是完全可以的,但酶切15分钟通常已经足够酶切充分了。
- 酶切完成后建议立即进行凝胶电泳分析,如果不能立即进行凝胶电泳分析,推荐加入1μL 0.1mM EDTA后-20℃保存。也可以80℃加热15min以失活T7EI酶后-20℃保存。
- 在变性和退火完成后,每管加入1μL的T7EI。
- 凝胶电泳分析:
- T7EI酶切反应结束后,向反应体系中加入4μL 6×DNA上样缓冲液,然后使用2%琼脂糖凝胶进行电泳分析。
- 使用凝胶成像系统拍摄电泳图像,并使用凝胶分析软件确定每个条带中所含不同长度的DNA的相对比例。
- 基因编辑效率的计算:
- 对于基因编辑产生随机突变的情况:
剪切比例(Fraction Cleaved) =被剪切条带的灰度总和(Sum of cleaved band intensities)/剪切条带和未被剪切的特异性条带的灰度总和(Sum of cleaved band intensities and specific parental band intensities)
Fraction Cleaved = (% gene modification)×(1 -% gene modification) + (% gene modification),求解可得:
基因编辑效率(% gene modification) = 100×(1 - (1 - Fraction Cleaved)1/2)
例如,当Fraction Cleaved = 75%时(即剪切条带的灰度总和为剪切条带和未被剪切的特异性条带的灰度总和的75%时),基因编辑效率= 50%。即当基因编辑效率为50%时,理论上观察到的剪切条带的灰度总和占所有特异性条带灰度总和的约75%。因为基因编辑发生时,例如CRISPR/Cas9,不会仅产生单一的一种基因编辑,而是会产生多种不同的插入或缺失突变,各种不同的插入和突变缺失体之间也能退火形成不完全匹配的双链DNA,并且能被T7EI所剪切的。 - 对于基因编辑仅产生特定突变的情况:
剪切比例(Fraction Cleaved) =被剪切条带的灰度总和(Sum of cleaved band intensities)/剪切条带和未被剪切的特异性条带的灰度总和(Sum of cleaved band intensities and specific parental band intensities)
Fraction Cleaved = (% gene modification)×(1 -% gene modification)×2,求解可得:
基因编辑效率(% gene modification) = 100×(0.5 ± 0.5×(1~2×Fraction Cleaved)1/2)
例如,当Fraction Cleaved = 37.5%时(即剪切条带的灰度总和为剪切条带和未被剪切的特异性条带的灰度总和的37.5%时),基因编辑效率= 25%或75%。即当基因编辑效率为25%或75%时,理论上观察到的剪切条带的灰度总和占所有特异性条带灰度总和的约37.5%。
- 对于基因编辑产生随机突变的情况:
- T7EI酶切反应结束后,向反应体系中加入4μL 6×DNA上样缓冲液,然后使用2%琼脂糖凝胶进行电泳分析。

- PCR扩增问题:
- 样品PCR扩增后无条带:如果PCR后样品管检测不到目的条带,而使用试剂盒提供的Control Templates & Primers能扩增出预期的条带,则可能需要重新设计引物,或调整退火温度。
- 拖带现象:细胞或组织样品裂解后提取的基因组DNA浓度过高,可尝试将基因组DNA稀释3~10倍后再进行PCR反应。
- 条带太弱:细胞裂解后提取的基因组DNA浓度过低,可尝试将PCR反应中基因组DNA的用量加倍。
注:请勿在PCR反应中使用超过2μL的裂解液,因为过多的裂解液会抑制PCR反应。
- 样品PCR扩增后无条带:如果PCR后样品管检测不到目的条带,而使用试剂盒提供的Control Templates & Primers能扩增出预期的条带,则可能需要重新设计引物,或调整退火温度。
- T7EI酶切问题:
- 样品条带没有被酶切:如果使用试剂盒提供的阳性对照Control Templates & Primers的扩增产物可以被T7EI酶切并产生剪切条带,而所需检测的样品没有检测到目的条带被剪切,可能由如下原因导致:
- 样品基因确实并没有被成功编辑,或者编辑的效率太低了。可以考虑尝试重新进行基因编辑(包括对于CRISPR/Cas9重新设计guide RNA,设法提高转染或感染效率。
- PCR引物离酶切位点过近。可以尝试重新设计距离酶切靶点更远的引物。
- 遗漏了变性和退火步骤。此时阳性对照Control Templates & Primers的扩增产物也是不能被酶切的。
- 样品基因确实并没有被成功编辑,或者编辑的效率太低了。可以考虑尝试重新进行基因编辑(包括对于CRISPR/Cas9重新设计guide RNA,设法提高转染或感染效率。
- 酶切产生的条带特别弱:可能需要重新设计或优化基因组编辑实验的条件,以提高基因编辑效率。也可以考虑后续筛选多克隆或单克隆细胞,以确保大部分细胞或所有细胞都发生基因编辑,便于用于后续的实验。
- 酶切产生了非特异性条带:
- 酶切时间过长或者酶过量。需要设置未进行基因编辑的阴性对照以区分背景和特异性剪切产生的条带。
- 酶切位点附近产生了非预期的突变,需要重新设计引物,以适当避免相应的非预期的突变位点。
- 酶切时间过长或者酶过量。需要设置未进行基因编辑的阴性对照以区分背景和特异性剪切产生的条带。
- 样品条带没有被酶切:如果使用试剂盒提供的阳性对照Control Templates & Primers的扩增产物可以被T7EI酶切并产生剪切条带,而所需检测的样品没有检测到目的条带被剪切,可能由如下原因导致:
相关搜索:基因编辑突变检测试剂盒,T7EI,T7核酸内切酶I,Gene-Editing Mutation Detection Kit



